
许多蛋白质都有糖链修饰,特别是在真核细胞的分泌蛋白中糖基化更是常见。糖基化可以促进蛋白质的质量控制或者介导蛋白质-蛋白质或细胞-细胞相互作用。
然而,这些糖基修饰通常十分复杂且多样,使用传统结构生物学或生物物理方法,很难对其进行深入的研究。
近日,Anggara 等人的研究表明,可以使用单分子原子力显微镜(STM)技术,直接对附着在多肽和脂质上的聚糖进行成像。这些生物大分可以通过温和的电喷雾技术,沉积到特制表面上;如果必要的话,还可以通过操纵来拉伸其结构区域。在该工作中,作者成功观察到了不同的聚糖结构和成像的大片段蛋白质,包括密集糖基化粘蛋白。

聚糖修饰的蛋白质和脂质是一种存在于所有生命体的现象,在生物功能和功能障碍中都起着重要作用。目前,目前,对这些被糖缀合物修饰的生物分子 (称为糖缀合物) 的分析策略,仍然依赖于整体平均的方法,即通过化学标记、化学酶消化和集成平均方法的组合来间接获得样品中最可能存在的结构。
但是,这种方法不能提供给定分子中附着在单个位点的聚糖的位置和结构的完整视图,特别是对于糖蛋白,因为结构异质和异构糖缀合物的整体平均分析模糊了与生物分子结合的聚糖的位置和结构,特别是对于具有多个聚糖连接的蛋白质。
因此,对单个分子的结构的见解丢失了集成平均分析,这阻碍了糖缀合物的结构-性质关系的研究。为了防止单个分子结构信息的丢失,需要在单分子水平上分析糖缀合物分子。
对聚糖修饰的单分子分子的困难,主要来自于糖缀合物所具有广泛的结构异质性 (即序列的多种变体)和结构异构性 (即具有相同质量的结构)。本文作者通过使用低温扫描隧道显微镜,完成了对单个糖缀合物分子进行直接成像来展示糖缀合物的单分子分析。
在作者建立的实验流程中,来自电喷雾的完整糖缀合离子软着陆在观察表面上,然后便可进行直接的单分子成像。量子力学模型证实的亚分子成像分辨率揭示了糖肽类、糖脂类、N-糖蛋白和 O-糖蛋白中密集修饰聚糖的整体结构和附着位点。
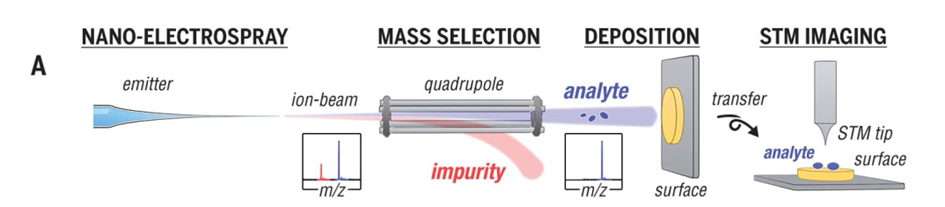
实验示意图: 大量选择 nESI 生成的糖缀合物或 GAG 离子,在 120 K 的低温下完整地软着陆在观察表面上,并在 11 K 的超低温下用 STM 进行成像(见方法)。
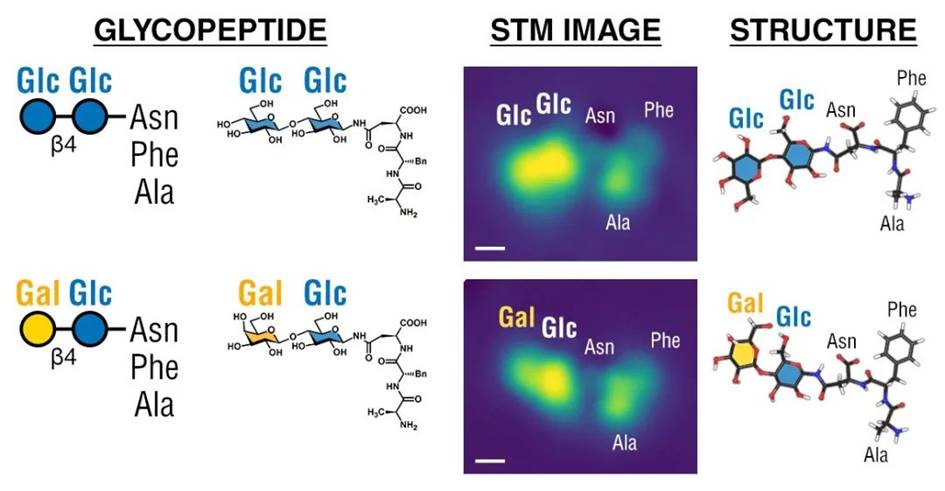
在建立了工作流程之后,作者首先用较为简单的糖基修饰类型进行了概念验证。作者从简单的糖基多肽(glycopeptide)开始,证明了该技术不仅可以区分聚糖和多肽,甚至还可以区分葡萄糖和半乳糖,而它们只有一个立体构型的不同,即-OH在C4上的不同!
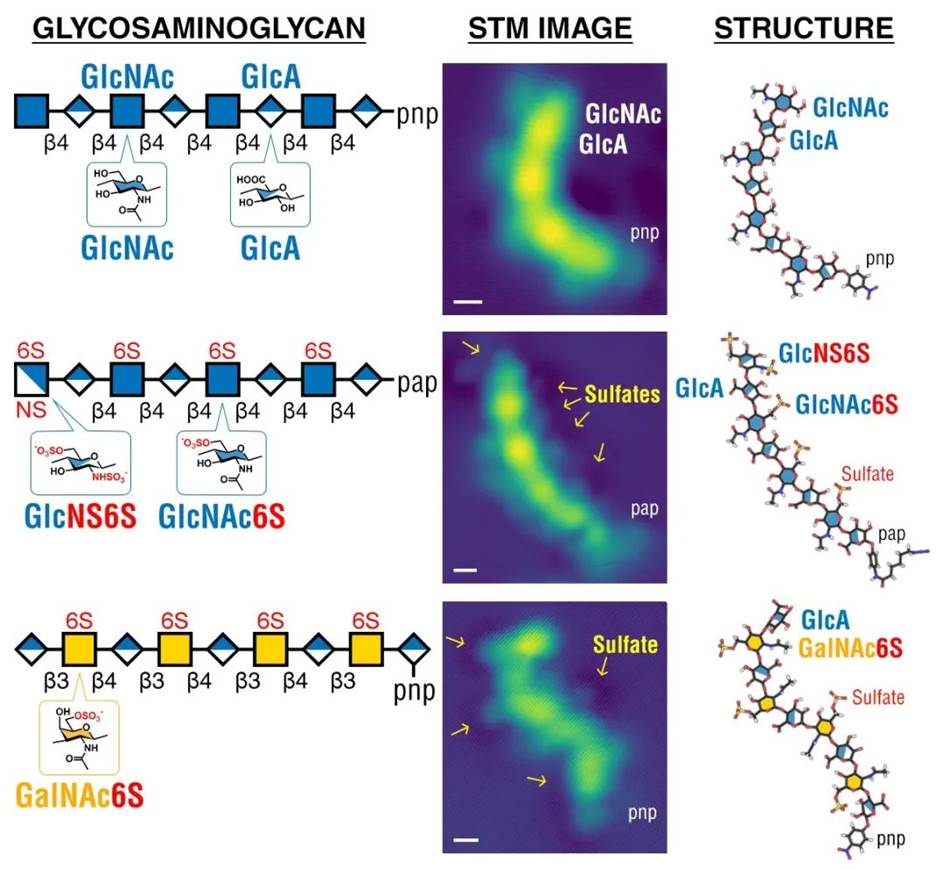
接下来,作者尝试用这套工作流程来看一些更复杂的东西,即糖胺聚糖 (又名GAGs)! 令人非常兴奋的是,作者发现该技术能够帮助人们找到 N-乙酰基和硫酸盐! 就此,作者猜想未来或许可以使用 STM 成像对“真正的”GAG 长链进行测序!
在上面研究的基础上,作者开始尝试研究更复杂的糖肽和糖脂!下面是一个唾液糖肽,以及 GM3 和 GD3 神经节苷脂,都提供作者建立的技术实现了单分子成像!
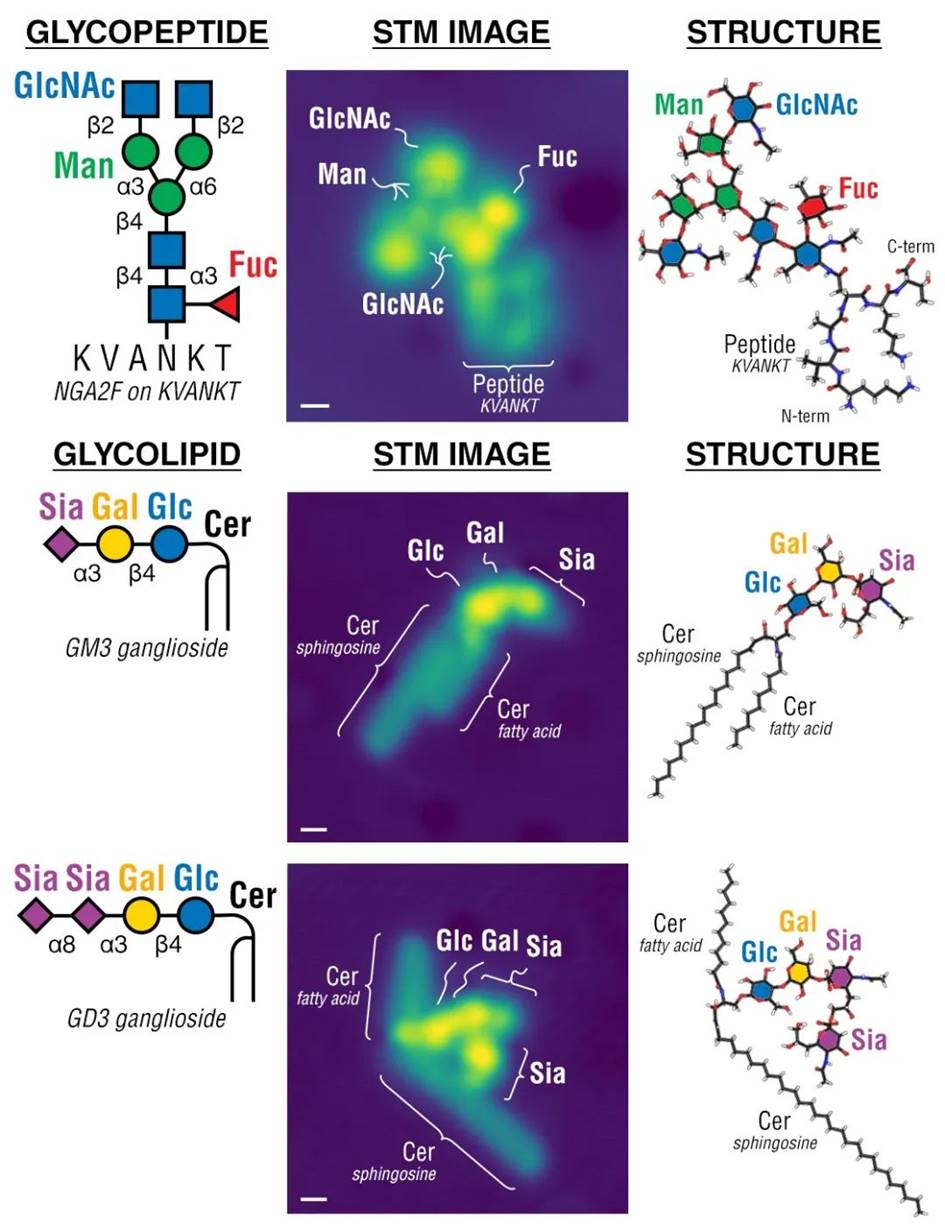
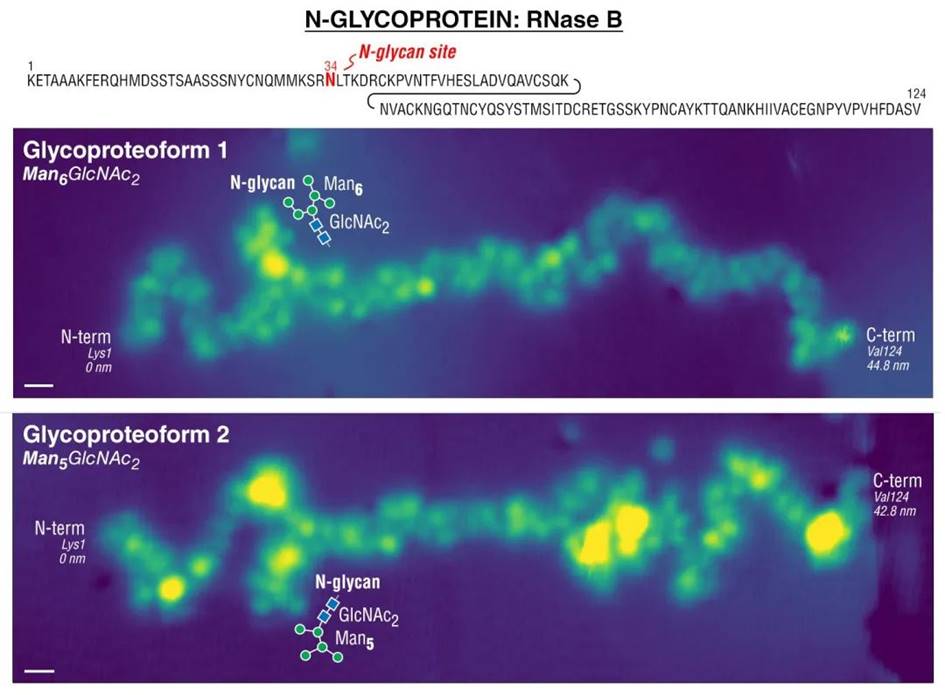
接下来就是糖蛋白了!单分子图像甚至能提供糖基化修饰的构型信息,也就是说可以帮助我们我们在单分子水平上区分糖型!也就是说,STM 的图像既可以告诉我们聚糖在蛋白质主干上的连接位置,也可以告诉那个位置的聚糖结构! 在 RNase B的图像中,N-糖基化分子的主链结构和走向都清晰可见!
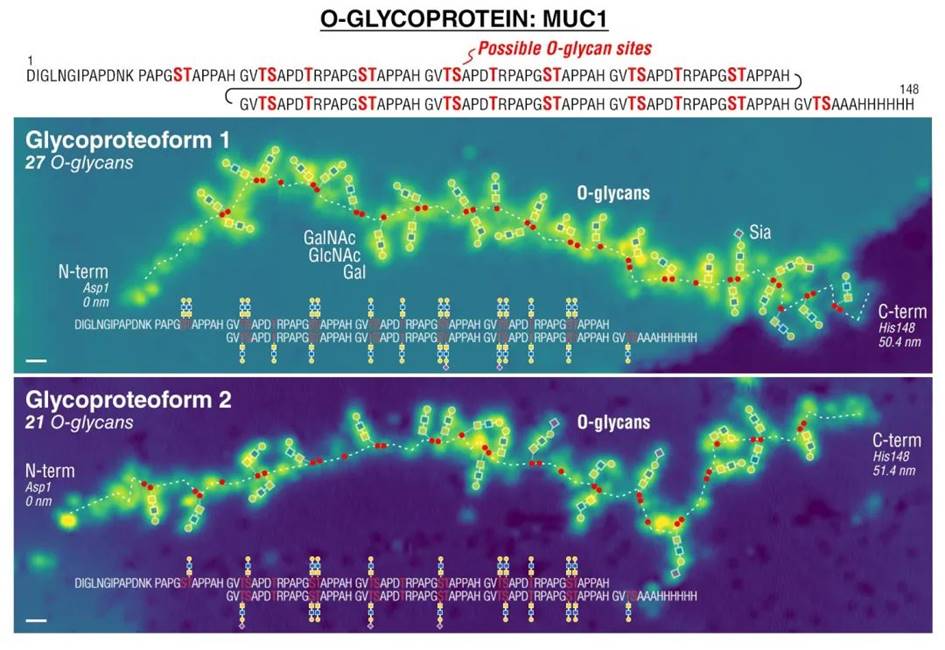
最后,我们来看看人类黏液蛋白 MUC1 的一个片段上所有的O型聚糖。MUC1报告基因 (148 AA) 的序列片段,包含20个氨基酸(GVTSAPDTRPAPGSTAPPAH) 的6.5个串联重复序列,这些氨基酸在 Ser (S) 和Thr (T) 残基上被 O-聚糖修饰 (总共有 34个潜在的 O-糖基化位点)。
总之,本文通过对单个糖缀合物分子进行直接、无标记、空间成像,实现了糖缀合物的单分子分析。作者展示了在亚纳米分辨率下对单糖缀合物进行成像,通过揭示其组成氨基酸、脂质和单糖亚基如何相互连接,揭示了每个分子的主要结构。因此,本文的成像方法通过定位糖缀合物分子中的每个单糖,根据其立体构型和侧基进行区分,从而在糖缀合物的每个聚糖附着位点上建立聚糖序列。作者展示了该方法用于广泛的糖缀合物,从简单的糖肽和糖脂到具有超过20个附加聚糖的复杂糖蛋白。本文的工作表明,单分子成像可以在单分子水平上直接获取与复杂的糖肽、糖脂和糖蛋白结合的所有聚糖的结构信息。
参考文献:Kelvin Anggara et al. ,Direct observation of glycans bonded to proteins and lipids at the single-molecule level.Science382,219-223(2023).DOI:10.1126/science.adh3856













